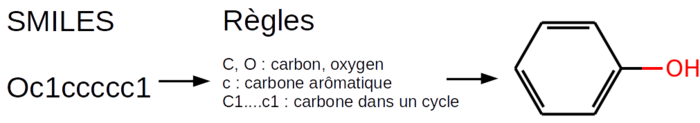La chimie est abordée bien avant le baccalauréat, mais l'introduction qui en est faite ne permet pas de mesurer l'importance que ces ensembles d'atomes très petits ont sur notre vie quotidienne. Nous allons passer en revue quelques rappels de chimie puis détailler les problèmes qui se posent rapidement pour le traitement de molécules virtuelles par des approches informatiques, ce que l'on pourrait résumer rapidement à une introduction à la chémoinformatique.
1. Représentation des molécules chimiques et complexité algorithmique
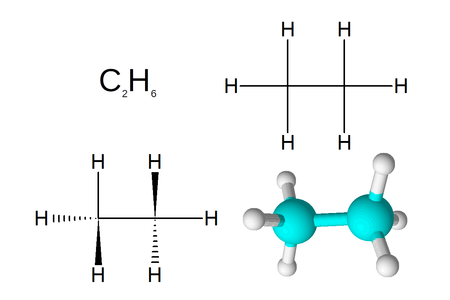
Si la table périodique des éléments comporte près de 120 types d'atomes stables (naturels ou synthétisés par l'homme), la nature n'exploite qu'une partie d'entre eux. En utilisant les atomes de carbone (C), d'oxygène (O), d'azote (N), et d'hydrogène (H) et dans une moindre mesure le phosphore (P) dans l'ADN et le soufre (S) dans les protéines, nous avons déjà la plupart des briques qui constituent les briques du vivant et les grandes classes de molécules chimiques : acides, bases, alcools, esters, aldéhydes, sulfates, etc. Pour représenter ces molécules, plusieurs notations permettent de retrouver rapidement la composition et l'organisation des molécules. La première, appelée aussi formule chimique brute, consiste à indiquer de manière séquentielle les atomes, le cas échéant avec des indices pour spécifier leur nombre, par exemple pour l'eau H2O (Figure 1). Cette écriture permet d'indiquer la composition d'une molécule, mais ne donne pas d'information directe sur les liaisons reliant les différents atomes. Il faut ainsi alors utiliser la formule développée plane pour apporter cette information. Il est ainsi possible de décrire si une liaison est simple, double ou triple par exemple dans le cas du carbone.
Les choses se compliquent lorsqu'il faut représenter de manière un peu plus réaliste les molécules qui sont en réalité en trois dimensions. Un simple atome de carbone lié à 4 hydrogène doit par exemple être représenté en deux dimensions pour faire apparaître des liaisons qui ont un angle de 109,47 degrés, dans une géométrie moléculaire tétraédrique. Pour répondre à ces problématiques, la représentation de Cram permet de représenter les molécules avec un trait si la liaison est dans le plan, par un triangle ouvert plein si la liaison est au-dessus du plan, ou par un triangle ouvert barré si la liaison est derrière le plan. Cette simple problématique de représentation projetée en 2D demande déjà une petite gymnastique en informatique pour respecter les nomenclatures officielles de l'organisme en charge de la normalisation, l'IUPAC [1].
Les choses se compliquent encore quand il s'agit de représenter réellement ces molécules en 3D, car de la formule brute peut découler plusieurs structures tridimensionnelles. Il suffit par exemple qu'un carbone dans une molécule ait tous ses atomes liés pour avoir deux possibilités d'organisation tridimensionnelle, on parle alors de carbone asymétrique. Dans cette situation cette molécule a deux configurations différentes appelées isomères (stéréo, diastéréo, énantiomères en fonction des situations). Sans rentrer dans le détail de toutes les possibilités rencontrées, cela implique qu'en plus des règles « simples » identifiées ci-dessus, il faut tenir compte de ces aspects pour générer une molécule en trois dimensions, ce qui augmente fortement la complexité algorithmique.
Fig. 1 : Rappels sur l'écriture de molécules chimiques et leur représentation, exemple de l'éthane. La formule brute en haut à gauche donne la composition chimique de la molécule, la formule développée donne une représentation éclatée de la molécule. Avec la représentation de Cram (en bas à gauche), il est possible de mieux prendre en compte l'organisation tridimensionnelle de la molécule, mais ce n'est qu'une fois disponible en trois dimensions, en représentation boules et bâtons, que l'on peut se rendre compte de la géométrie réelle des molécules, avec les carbones en bleu et l’hydrogène en blanc.
Après ce bref rappel sur les notions classiques d'écriture « à la main » de molécules chimiques, nous allons pouvoir détailler ce qui permet de manipuler virtuellement des molécules.
2. Représentation virtuelle des molécules
Même si l'objectif est de représenter en 3D les molécules chimiques (nous verrons plus loin pourquoi), pour des raisons de performances et de facilité de manipulation, il est nécessaire d'avoir recours à des représentations linéaires (1D) et dans le plan (2D) comme présenté dans la figure 2. La notation la plus classique en mode 1D est le format SMILES. Ce format, inventé par le fondateur de l'entreprise DAYLIGHT permet de décrire de manière linéaire l'organisation d'une molécule en 3D (ou pseudo-3D) sur une seule ligne [2]. Les atomes sont représentés par une lettre majuscule, les liaisons multiples sont indiquées explicitement, et les atomes impliqués dans un cycle aromatique sont en minuscule. Comme la position des hydrogènes peut être déduite de la position des atomes lourds, ils ne sont pas indiqués.
Hormis ces conventions de nommage, le fait important pour notre analyse est que cette représentation fait appel à la théorie des graphes, graphes pour lesquels les arêtes sont les liaisons qui relient les sommets du graphe : les atomes. Cette utilisation de la théorie des graphes est assez ancienne (déjà dans les années 1970 …), et en fonction des sources, peut probablement être retrouvée avant la formalisation même de la théorie moderne sur les graphes.
Fig. 2 : Illustration du passage de la notation du format SMILES au format 2D à l'aide d'un jeu représentatif de règles.
La possibilité de représenter les molécules sous forme de graphe va permettre d'exploiter les nombreuses possibilités de cette théorie. Nous n'allons pas développer les concepts sous-jacents pour chacun des aspects de la théorie des graphes, mais simplement profiter du fait que l'analyse d'une molécule chimique sous forme de graphe permet de faire abstraction d'une partie des contraintes et limitations vues précédemment. Passons maintenant à la réalisation concrète de modélisation de molécules et à l'utilisation de méthodes associées.
3. Création d'une petite chimiothèque
L'objectif de la chémoinformatique est d'étudier à grande échelle un ensemble de molécules chimiques pour étudier leurs caractéristiques intrinsèques (est-ce que les molécules sont proches ou non ?), ou pour prédire leur comportement sur une cible biologique donnée. Quand un médicament est connu pour se fixer à une protéine du corps humain (pour nous soigner), il est très important et intéressant de trouver toutes les molécules qui pourraient potentiellement ressembler à la molécule de départ, pour diminuer les effets secondaires par exemple. Pour illustrer comment il est possible de faire à petite échelle ces analyses, nous allons faire appel à la bibliothèque RDKit qui connaît un essor important ces dernières années [3].
Les logiciels disponibles dans RDKit sont écrits en C++, ils sont donc rapides et performants. Fort heureusement une traduction des fonctions est réalisée dans différents langages (Java, C# grâce à Swig), mais nous nous contenterons de Python. Les étapes d'installation ne seront pas détaillées, car plusieurs modalités sont accessibles, de la compilation à partir des sources à l'utilisation de Conda pour tout automatiser. Attention au temps d'installation qui peut prendre quelques heures… Pour faciliter la reprise des explications de l'article, nous allons utiliser iPython et la console qt pour l'affichage des images. Les étapes détaillées ci-après seront disponibles sous forme de notebook Jupyter [4].
Pour enregistrer notre travail, nous allons utiliser la version qt de IPython qui permet l'affichage en direct des molécules, et de matplotlib pour générer quelques images. L'article a été rédigé sous Ubuntu 14.04, c'est donc Python 2.7 qui est la version par défaut.
$ sudo apt-get install ipython-qtconsole python-matplotlib
$ ipython qtconsole
Dans le terminal IPython qui s'ouvre, nous allons indiquer au système où se trouve l'installation de rkdit :
>>> importsys
>>>sys.path.append('/opt/rdkit-Release_2016_09_4/')
Il faut ensuite importer les modules cœur qui vont permettre d'effectuer la création et la manipulation virtuelle de molécules :
>>> from rdkit import Chem
>>> from rdkit.Chem.Draw import IPythonConsole
>>> from rdkit.Chem import Draw
Notez au passage qu'il est possible d'avoir plusieurs lignes dans iPython sans avoir la notification d'une nouvelle attente qui commence par la ligne In[numéro].
Il est maintenant possible de créer votre toute première molécule virtuelle, nous allons commencer par le phénol. Comme la notation SMILES n'est pas simple à créer à partir de zéro, nous allons nous rendre sur un site de référence pour les composés à activité biologique : ChEBI.
Une courte recherche sur le terme « phenol » permet d'identifier la molécule du même nom, et en cliquant sur la fiche dédiée, le code SMILES est fourni : Oc1ccccc1. Nous allons créer l'objet correspondant dans RDKit.
>>> phenol=Chem.MolFromSmiles('Oc1ccccc1')
Il suffit d'appeler l'objet sans paramètre pour obtenir un dessin de celui-ci dans le notebook.
>>> phenol
Nous allons y ajouter les numéros d'atomes pour faciliter la comparaison entre plusieurs structures par la suite.
>>> from rdkit.Chem.Draw.MolDrawing import DrawingOptions
>>> DrawingOptions.includeAtomNumbers=True
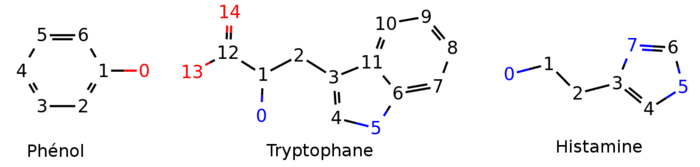
Deux autres molécules sont ajoutées à la session du notebook, le tryptophane et l'histamine, en procédant de la même manière qu'au point 4 : recherche sur ChEBI, création de l'objet dans RDKit, et affichage. Le notebook contient maintenant trois molécules.
>>> trp = Chem.MolFromSmiles('NC(Cc1c[nH]c2ccccc12)C(O)=O')
>>> histamine = Chem.MolFromSmiles('NCCc1c[nH]cn1')
>>> my_molecules =[phenol,trp,histamine]
>>> legends =['Phenol','Tryptophane','Histamine']
>>> Draw.MolsToGridImage(my_molecules,legends=legends)
Le résultat de ces commandes est disponible dans la figure 3.
Fig. 3 : Image dans le plan du phénol, du tryptophane et de l'histamine une fois créés à partir de leur séquence SMILES dans RDKit. La grille permet de visualiser rapidement la composition d'une chimiothèque pour rapidement identifier les molécules qui la composent.
4. Étude de la chimiothèque
Les molécules intégrées dans le notebook ne se ressemblent pas parfaitement, il faut donc trouver un critère qui va permettre de les comparer. Une approche naïve consisterait à chercher à identifier les atomes identiques entre les différentes molécules, à les aligner puis à compter le nombre d'atomes ainsi mis en correspondance. Cette approche pourrait fonctionner à petite échelle, mais comme nous l'avons vu précédemment, les molécules chimiques peuvent être très proches au niveau de la formule brute, et très différentes une fois le problème des configurations pris en compte. Une autre approche serait de chercher à les superposer « visuellement », mais il risque d'être difficile de trouver une correspondance satisfaisante. Ces deux approches ne peuvent pas être retenues : elles sont source d'erreurs importantes, et même en cas de succès, le temps de calcul (ou humain) serait prohibitif.
C'est maintenant l'occasion de revenir sur une propriété fondamentale décrite ci-dessus : il est possible de représenter les molécules comme des graphes. Cette approche permet de résoudre beaucoup de problèmes en une fois : il n'y a pas besoin de définir l'organisation en deux ou trois dimensions de la molécule, car l'information est contenue dans le graphe, il n'y a plus de dépendance quant à l'orientation des molécules entre elles, car les graphes ne dépendent pas de ces propriétés. L'approche qui a été développée consiste à étudier la molécule comme un sous-ensemble de chemins potentiels, et à stocker ces chemins dans un vecteur linéaire, par défaut de 1024 bits. Par exemple, chaque chemin constitué de 7 atomes va être isolé comme un bit dans le vecteur de description, et l'ensemble des bits (activés) ou non va être nommé « empreinte » ou fingerprint en anglais.
Il existe plusieurs algorithmes pour généraliser l'approche de chemins de 7 atomes, l'approche la plus efficace à ce jour est basée sur l'algorithme ECFP, celle utilisée par défaut dans RDKit est l'empreinte de Morgan. Pour plus de détails sur ces algorithmes et une explication plus poussée, le lecteur pourra consulter les liens fournis en fin d'article [6].
Nous allons visualiser le résultat de ces comparaisons pour déterminer quelle molécule est plus proche du phénol (la plus petite molécule de notre jeu de données).
>>> from rdkit.Chem.Draw import SimilarityMaps
>>> SimilarityMaps.GetSimilarityMapForFingerprint(phenol, trp, SimilarityMaps.GetMorganFingerprint)
>>> SimilarityMaps.GetSimilarityMapForFingerprint(phenol, histamine, SimilarityMaps.GetMorganFingerprint)
Le résultat de la comparaison des molécules deux à deux est présenté dans la figure 4.
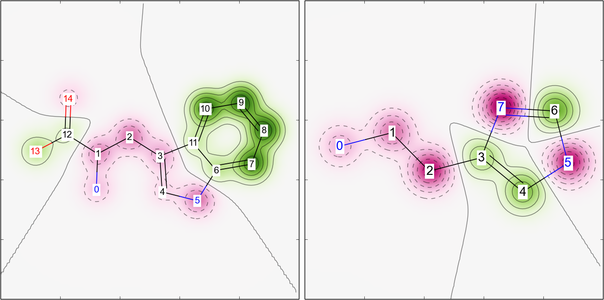
Fig. 4 : Comparaison des empreintes entre différentes molécules à l'aide d'empreintes de Morgan. À gauche, illustration du résultat des correspondances identifiées entre le phénol et le tryptophane, à droite le résultat de la comparaison entre le phénol et l'histamine. La coloration en vert des atomes indique qu'ils participent à la complémentarité entre les molécules, la coloration en rose indique les zones de divergence. Quand le rôle d'un atome est neutre dans la comparaison des molécules, il est indiqué en gris.
Cette analyse qualitative montre la difficulté de réaliser cette opération à la main, la correspondance entre les atomes ne pouvant être exacte, seules certaines parties du phénol sont prises en compte pour la superposition. Pour mieux caractériser les interactions, il faut utiliser le vecteur de l'empreinte, et comparer les vecteurs deux à deux. La valeur numérique obtenue s'appelle le critère de Tanimoto, du nom de son inventeur, plus la valeur est proche de 1 et meilleure est la correspondance.
>>> from rdkit.Chem import AllChem
>>> from rdkit import DataStructs
>>> fp_query = AllChem.GetMorganFingerprintAsBitVect(phenol,2)
>>> fp_phenol = AllChem.GetMorganFingerprintAsBitVect(phenol,2)
>>> fp_trp = AllChem.GetMorganFingerprintAsBitVect(trp,2)
>>> fp_histamine = AllChem.GetMorganFingerprintAsBitVect(histamine,2)
>>> DataStructs.FingerprintSimilarity(fp_query, fp_phenol)
1.0
>>> DataStructs.FingerprintSimilarity(fp_query, fp_trp)
0.19444444444444445
>>> DataStructs.FingerprintSimilarity(fp_query, fp_histamine)
0.06666666666666667
Cette analyse nous indique, fort heureusement, que le phénol est bien le phénol, et que sa proximité avec le tryptophane n'est que de 0.19, et est encore plus éloignée pour l'histamine (0.07). La valeur entre l'histamine et le tryptophane est triviale à trouver, elle est laissée à la discrétion du lecteur…
Conclusion
Nous avons vu brièvement lors de cet article qu'il est essentiel de lier la représentation de petites molécules avec des notions informatiques et algorithmiques complexes. Pour la démonstration, seule une petite chimiothèque a été créée, la norme dans ce domaine est plutôt de l'ordre de millions ou de dizaines de millions de molécules. L'intérêt de pouvoir représenter des molécules sous forme de graphe est multiple. Il permet tout d'abord d'utiliser des métriques établies, il permet ensuite de classer une chimiothèque pour définir des molécules par proximité, et il présente un temps d'exécution très court.
Une fois que la chimiothèque a été traitée, puisque les informations sont numériques, il est possible d'utiliser des approches d'apprentissage avancé. Par exemple, en couplant RDKit et scikit-learn [7], il est possible de faire le lien entre une empreinte et une activité biologique. Vous pouvez parcourir l'article scientifique référencé en annexe (au moins pour les titres des méthodes) pour voir l'exploitation du machine learning faite dans ce domaine [8]. Les approches par réseaux de neurones sont déjà couramment utilisées, reste à savoir si les dernières évolutions dans le deep learning vont permettre de concevoir de nouvelles molécules thérapeutiques, meilleures que celles existantes.
Références
[1] Conférence internationale sur les molécules chimiques : https://iupac.org/
[2] Les règles simplifiées de représentation des molécules avec l'encodage SMILES : http://www.daylight.com/dayhtml/doc/theory/theory.smiles.html
[3] Rdkit, le couteau suisse de la chémoinformatique : http://www.rdkit.org/
[4] Le notebook iPython, Jupyter : https://jupyter.org/
[5] Les substances chimiques d'intérêt biologique : https://www.ebi.ac.uk/chebi/
[6] Explication détaillée de la construction de l'empreinte ECFP : https://docs.chemaxon.com/display/docs/Extended+Connectivity+Fingerprint+ECFP
[7] Une des bibliothèques d'apprentissage en Python : http://scikit-learn.org
[8] Inventaire des méthodes d'apprentissage pour le traitement de chimiothèques : https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4180928/



 Ajouter à une liste de lecture
Ajouter à une liste de lecture