Les protéines sont des composants biologiques importants constitués par l'assemblage de briques élémentaires, les acides aminés. L'organisation tridimensionnelle d'une protéine est cependant difficile à prédire à partir de la succession d'acides aminés. Dans cet article, nous allons détailler une étape importante dans le processus de construction d'un modèle tridimensionnel, la prédiction d'éléments de structure secondaire remarquables.
Avec l'avancée phénoménale des moyens informatiques et des technologies de séquençage de l'ADN, il est possible de séquencer l'ensemble du génome d'une personne en moins d'une semaine. Cette séquence brute d'ADN peut ensuite être traitée pour identifier les gènes spécifiques qui vont donner différentes protéines. Pour un génome humain par exemple, cela représente environ 20000 gènes, qui vont conduire à la production dans les différentes cellules de notre corps de une à dix protéines par gène.
À l'heure actuelle, il est très difficile d'obtenir une structure tridimensionnelle d'une protéine, il est donc nécessaire d'utiliser de nombreuses approches de prédictions de motifs remarquables pour contraindre les degrés de liberté à explorer pour la production d'un modèle 3D raisonnable. Nous allons nous intéresser dans cet article à un aspect de ce processus de prédiction, la prédiction des éléments réguliers de structure secondaire.
1. Composition des protéines et difficultés de prédiction de la structure tridimensionnelle
Une protéine est composée de 20 acides aminés, qui, de par leurs caractéristiques physico-chimiques spécifiques, vont conduire à un repliement spontané en une structure tridimensionnelle unique [1]. Il n'est pas possible d'explorer par la force brute toutes les combinaisons de repliement d'une protéine. Compte-tenu du nombre de degrés de liberté à prendre en compte, le temps nécessaire dépasserait l'âge même de l'univers. Ce paradoxe a été écrit pour la première fois par Levinthal en 1969 [2].
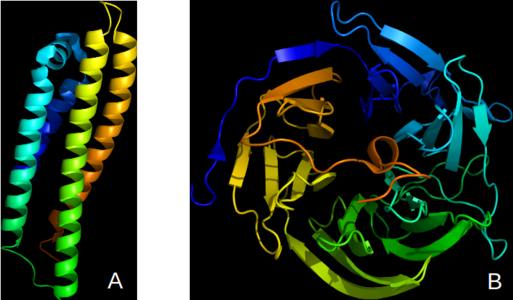
La nature arrive pourtant à obtenir un repliement stable dans une période beaucoup plus courte, entre la microseconde et la dizaine de secondes. Pour reproduire ce comportement, il faut diminuer les degrés de liberté à explorer en faisant appel à la prédiction de caractéristiques macroscopiques bien définies. Quelle que soit la taille de la protéine, il est possible de classer les éléments remarquables en trois grandes catégories : les hélices alpha, les feuillets bêta, et les segments non structurés (voir figure 1).
Fig. 1 : Exemple de structures secondaires remarquables dans les protéines, coloriées en arc-en-ciel du N-terminal au C-terminal de la séquence protéique. (A), la protéine apolipoprotéine E, qui intervient dans le métabolisme des lipides et tout particulièrement du cholestérol, possède uniquement des hélices alpha. (B), la diisopropylfluorophosphatase de calmar a une fonction inconnue, mais sa structure présente principalement des feuillets bêta, organisés en 5 motifs répétés rappelant une hélice de bateau.
De nombreux travaux ont pour objectif de répertorier les structures tridimensionnelles connues ; ils sont regroupés dans la Protein Data Bank qui vient de fêter son 46ème anniversaire [3]. Plus de 130 000 structures de macromolécules sont ainsi identifiées à ce jour. En analysant chacune de ces structures, il est possible de classer les protéines selon leurs éléments de structures secondaires, comme permet de le faire la base de données CATH (Class, Architecture, Topology, Homology) [4]. Ces éléments de structure secondaire sont dans un premier temps imaginés comme étant dans la conformation « idéale », mais dans la pratique de nombreuses déformations sont observées (courbure, torsion, etc.), ce qui rend leur détection moins triviale [5]. Fort heureusement, il existe une méthode standard pour attribuer ces éléments de structure secondaire sur une structure tridimensionnelle existante, DSSP, que nous ne détaillerons pas dans le cadre de cet article [6].
Puisque les méthodes de détermination expérimentale existent, et qu'il est possible de caractériser les éléments de structure secondaire de manière détaillée, pourquoi détailler ces informations dans cet article ? Nous allons présenter les problèmes qui se posent et les solutions apportées juste après...
2. Approches méthodologiques pour la prédiction des éléments de structure secondaire
Les structures des protéines demandent de nombreuses années pour être déterminées, alors qu'à l'inverse, la caractérisation de la séquence de cette protéine (la suite de lettres qui indique sa composition en acides aminés) est très rapide. Cet écart entre les données tridimensionnelles et les données de séquence est visible dans les données publiques disponibles. Au moment de rédiger cet article, il y a ainsi plus de 90 millions de séquences de protéines, quand seulement plus de 130 000 sont disponibles en 3D… Ce gouffre entre les deux informations ne cesse de s'ouvrir, à la manière de la dorsale médio-atlantique, et ce n'est que par des moyens informatiques qu'il sera possible de le combler.
Comme nous l'avons vu précédemment, il est possible de réduire le nombre de degrés de liberté pour prédire la structure tridimensionnelle des protéines en identifiant des segments qui seront considérés comme un bloc indivisible, les éléments de structure secondaire. En ne considérant uniquement que les angles qui séparent deux acides aminés, phi et psi, et en considérant que chaque angle n'accepte qu'un nombre discret d'orientation (6, soit des pas de 60°, pour simplifier), cela revient à réduire le nombre de degrés d'un système de 8 acides aminés de 2 (8-6), soit 128 degrés de liberté ou encore 128*6*6 = 4608 combinaisons à évaluer à 3. En fonction de l'attribution d'une structure par le logiciel de prédiction, on utilisera en effet la géométrie idéale pour une hélice alpha, pour un feuillet bêta, ou il n'y aura pas de géométrie imposée. Pour rappel, une protéine a une taille moyenne de 300 acides aminés et les angles phi et psi ont une plage exploitable beaucoup plus importante que 6 valeurs chacun [7].
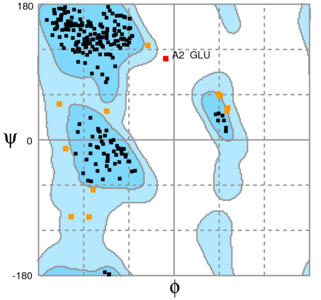
La figure 2 présente la répartition des angles phi et psi pour la protéine de calmar.
Fig. 2 : Détail des angles PSI et PHI identifiés dans la structure de l'enzyme de calmar. L'image a été obtenue à l'aide de l'outil RAMPAGE (http://mordred.bioc.cam.ac.uk/~rapper/rampage.php).
Les structures secondaires répondent ainsi à des règles moins combinatoires qu'une approche « naïve » (ou de force brute) ne pourrait le laisser penser. En résumé, il est possible de prédire pour une séquence donnée si pour un acide aminé donné il présente une structure secondaire remarquable, mais :
- la caractérisation de cet élément de structure secondaire sur les données disponibles n'est pas triviale ;
- le nombre de degrés de liberté à prendre en considération est important ;
- le jeu de données tridimensionnelles disponible est restreint (et biaisé).
Pour répondre à ces différents points, les premières approches ont cherché à déterminer de manière statistique lequel, parmi les 20 acides aminés, présentait statistiquement un biais de présence dans une structure secondaire ou une autre. En considérant une protéine idéale de 100 acides aminés, avec 20 acides aminés disponibles, la probabilité de chaque acide aminé est de 5 %. Pour savoir si un acide aminé était sous ou sur-représenté dans un état hélice, feuillet ou indéterminé (coil), il a suffi de déterminer la proportion de chacun des acides aminés dans les différentes structures. Cette approche a été initiée par Chou et Fasman dès 1974 [8]. Parmi les règles identifiées, il est possible de distinguer les acides aminés surreprésentés dans les structures en hélice alpha (alanine, aspartate, glutamine, leucine, methionine) ou à l'inverse ceux qui vont participer à la rupture d'une structure secondaire remarquable (glycine et proline). De nombreuses méthodes de prédiction, basées sur des évolutions de ces analyses préliminaires ont ainsi vu le jour, en particulier chez nos collègues francophones pour n'en citer que deux, GOR ou SOPMA [9].
Ces approches ont été basées sur une connaissance très partielle de la structure des protéines, au moment où ces analyses ont eu lieu, quelques dizaines à quelques centaines de structures tridimensionnelles (et donc de données que l'on pouvait dériver d'entre elles…) étaient disponibles. Avec l'évolution de la recherche, les nouvelles structures ont montré qu'il était nécessaire de faire appel à des stratégies de prédiction plus développées.
3. Remplacer l'humain avec du machine learning
Si l'analyse statistique des structures pouvait donner de grandes tendances entre la composition en acides aminés et un élément de structure secondaire, il est vite devenu nécessaire de prédire avec confiance les structures secondaires pour un ensemble de protéines dont la structure n'était pas connue.
3.1 PSIPRED, étape 1 : rechercher les séquences similaires
Parmi les approches évaluées par la communauté scientifique, c'est David Jones qui a apporté avec son outil PSIPRED une méthodologie de prédiction fiable et rapide. Il a ainsi exploité la toute nouvelle version de l'outil BLAST, qui sert à chercher les séquences proches d'une séquence d'intérêt dans un océan de séquences disponibles (un moteur de recherche de séquences, voir encadré pour plus de précisions). Cette version fait appel à un profil intermédiaire pour trouver de nouvelles séquences : une première recherche permet de trouver les séquences « proches » (comme un moteur de recherche) puis un profil est établi.
BLAST et BLOSUM
La recherche d'une séquence proche dans un océan de séquences disponibles est non triviale. De par leur nature évolutive, les séquences brutes en acides aminés d'une protéine vont vite diverger entre différents organismes, alors que la structure tridimensionnelle et la fonction de la protéine vont être beaucoup plus conservées. Pour avoir une analogie, il suffit de penser à une voiture. L'organisation générale comprend 4 roues, un moteur, un volant, des sièges, etc., mais dans le détail il existe beaucoup de types de voitures différentes.
L'algorithme Basic Local Alignment Search Tool (BLAST) utilise une approche heuristique (c'est-à-dire non exhaustive) : pour comparer deux séquences, il cherche dans un premier temps les segments courts (appelés k-mers) qui peuvent être alignés de manière exacte entre eux. Il cherche ensuite quelle est la combinaison de ces segments courts qui permet d'obtenir une reconnaissance idéale, appelée hit. Pour identifier ces segments courts identiques ou proches, appelés high scoring pairs (HSP), il fait appel à deux notions importantes. La première est le pourcentage d'identité de séquence, c'est-à-dire le nombre d'acides aminés pour un segment donné (ou toute la séquence) pour laquelle il existe une correspondance exacte. Si 4 acides aminés sur 5 dans un segment sont identiques, alors le pourcentage d'identité sera de 4/5 soit 80 %. Dans les cas où la correspondance n'est pas exacte, il faut prendre en compte une pénalité pour substituer un acide aminé pour un autre. Ces pénalités ont été établies sous forme de matrice de substitution sur un jeu de données de référence, la matrice la plus connue est nommée BLOSUM. Plus de détails sont disponibles dans les liens fournis en conclusion de l'article.
Ce profil permet de pondérer le résultat de la première recherche en donnant un poids plus important aux lettres qui reviennent le plus souvent. Si la séquence A contient « THECATHASFAT » et la séquence B contient « THECATISFAT » alors le profil P contiendra les lettres « THECATIS...FAT ». Ce profil établi, le logiciel va réutiliser non pas la séquence A pour la recherche, mais le profil obtenu, qui permet de mieux pondérer l'intérêt des acides aminés pour leur donner une priorité plus grande dans la seconde phase de la recherche. Ce processus itératif a été répété trois fois pour générer les données d'apprentissage de PSIPRED. Une illustration de ces profils est donnée dans la Figure 3.
Fig. 3 : Exemple de profil obtenu après recherche de séquences proches de la protéine B de calmar. Ce format appelé WebLogo (http://weblogo.berkeley.edu/logo.cgi) permet de visualiser rapidement les acides aminés conservés entre différentes espèces. À la position 234, la lysine (K) est conservée à 100 % dans toutes les séquences tandis qu'à la position 235 une sérine (S) et une asparagine (N) peuvent être observées avec une probabilité identique.
Cette approche itérative présente deux intérêts majeurs :
- Elle permet de mieux mettre en avant les éléments qui sont conservés localement, ce qui permet un lien direct avec les structures secondaires, car les éléments intermédiaires sont moins considérés ;
- Comme seule la première séquence A de l'alignement est celle pour laquelle il existe une structure tridimensionnelle et donc l'attribution d'éléments de structure secondaire, l'alignement local détecté avec PSIBLAST pour les acides aminés à la même position dans une autre séquence B, C, D, E, etc. permet de détecter quels acides aminés peuvent potentiellement remplacer celui original tout en conservant les éléments de structure secondaire.
Ces deux aspects sont très importants, car il est connu que la conservation d'un acide aminé est directement proportionnelle à son importance d'un point de vue de l'évolution. Comme nous l'avons vu dans la figure 1, en fonction de l'emplacement d'un acide aminé, celui-ci va être impliqué dans une hélice ou un feuillet, et sa position va varier (il peut être au début de l'élément, au milieu, ou à la fin). Le résultat d'une recherche dans PSIBLAST permet de détecter rapidement certains de ces acides aminés : s'il s'agit d'un résidu impliqué dans un rôle fonctionnel, par exemple pour une enzyme, alors il sera strictement conservé et retrouvé chez tous les animaux, et parfois même chez les bactéries. Pour certains acides aminés, une conservation plus faible est observable, mais toutefois significative, ces résidus ont le plus souvent un rôle dans la structure de la protéine, un peu comme si dans une maison on n'avait pas les dimensions de la fenêtre et des portes, mais que l'on savait qu'il fallait quatre fenêtres et deux portes...
3.2 PSIPRED, étape 2 : prédiction de l'état secondaire
Le jeu de données d'apprentissage est constitué de deux éléments :
- l'information sur l'état de structure secondaire (H, S, D pour Helix - Hélice, Sheet – Feuillet et Disordered - Désordonné respectivement) pour la protéine pour laquelle on dispose de la séquence et de la structure ;
- d'un alignement biaisé des séquences disponibles avec la séquence de la protéine (voir figure 4).
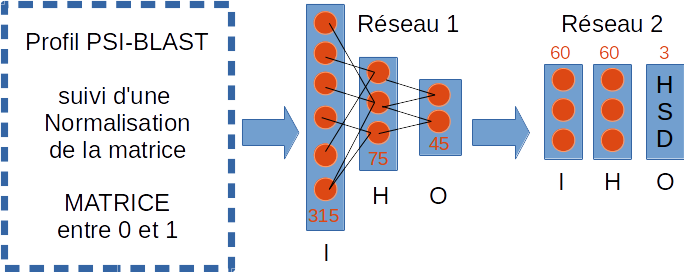
Fig. 4 : Schéma simplifié du fonctionnement de PSIPRED. À partir du profil établi avec PSI-BLAST, les possibilités de substitution d'une position pour un acide aminé donné sont transformées en vecteurs numériques en se basant sur les scores de substitution de BLOSUM62. Ces vecteurs sont ensuite utilisés en entrée (I) du premier réseau de neurones pour obtenir trois vecteurs correspondant aux états à modéliser. Celui-ci contient une couche cachée (H) composée de 75 neurones, et une couche de sortie (O) composée de 45 neurones. La sortie de ce premier réseau est utilisée et complétée par un vecteur spécifique pour les acides aminés aux extrémités dans un second réseau de neurones contenant 60 neurones dans la couche cachée et trois neurones dans la sortie définitive, qui donne la prédiction à trois états.
Un premier réseau de neurones est constitué en entrée de 315 entrées, d'une seule couche cachée contenant 75 neurones et de trois états en sortie (H,S, D) répartis chacun en vecteurs de 15 sorties. Les 315 entrées correspondent à un vecteur par acide aminé de 15 séquences issues de la recherche avec PSI-BLAST (15 séquences x 20 acides aminés), et d'un vecteur supplémentaire pour les acides aminés de début (N-terminal) et fin de séquence (C-terminal).
Un second réseau de neurones utilise la sortie du premier réseau de neurones pour déterminer l'état final à trois lettres. Cette fois-ci, il reprend les 3*15 vecteurs en entrée, en ajoutant un vecteur de 15 pour prendre en compte les extrémités de la séquence, ce qui fait au total 60 entrées. Une couche cachée unique est utilisée pour l'apprentissage, elle possède 60 neurones. La sortie se fait avec les trois états que l'on cherche à déterminer (H,S, D).
L'apprentissage a été réalisé avec un modèle simple de back-propagation et un taux d'apprentissage. Pour plus de détails sur ces paramètres et leur intérêt, veuillez vous reporter à une excellente source de documentation [10].
3.3 PSIPRED, étape 3 : validation et performance
Le jeu de prédiction et de test utilisé par l'auteur en 1999 était très réduit, il contenait 187 protéines et moins de 340000 séquences (le nombre exact n'est pas donné, l'auteur a filtré des séquences pour enlever de la redondance et du bruit). Cela implique qu'il était possible d'attribuer via une mesure (DSSP) les structures secondaires pour 187 séquences seulement... Ces séquences ont été séparées en trois jeux de données d'une taille de 62, 62 et 63 séquences. Pour la mise en place des réseaux, seuls 90 % des séquences de chaque jeu de données ont été utilisés pour éviter le phénomène de sur-apprentissage : si on donne toutes les données à un réseau, il arrive à « tout apprendre », mais on biaise ainsi le résultat.
L'article original mentionne un taux de prédiction sur des cibles connues, mais non utilisées pour l'apprentissage qui fluctue entre 75 et 80 %. Jusqu'à nos jours, c'est un taux de prédiction très important qui n'a pas été dépassé de manière robuste par les autres méthodes explorées.
Conclusion
Nous avons vu dans cet article un cas très intéressant de machine learning appliqué à un problème complexe en biologie. Dans toutes les situations à traiter par des méthodes d'apprentissage, il y a trois étapes à respecter :
- la constitution d'un jeu de données représentatif non redondant ;
- le choix d'une méthode d'apprentissage ;
- la validation de la prédiction sur un jeu de données externes.
Dans le cadre de cette méthode, l'objet à prendre en considération est complexe, avec des écarts très importants entre les informations mesurables (les structures secondaires sur quelques milliers de protéines) et les informations à prédire, à partir des séquences de millions de protéines.
Le travail effectué par David Jones en 1999 a fait date : c'était la première fois qu'une méthode rapide, « simple », permettait une prédiction robuste, quel que soit le sujet d'étude considéré, avec un taux de bonne prédiction variant de 75 à 80 %. Depuis cette époque, les jeux de données ont beaucoup évolué avec plus de 40000 protéines uniques pour lesquelles il existe une structure tridimensionnelle et plus de 90 millions de séquences pour lesquelles il faut une prédiction de structure secondaire …
De nombreuses approches ont été explorées pour mieux utiliser le jeu de données, en faisant appel à d'autres technologies comme le SVM, le deep learning, des statistiques avancées, etc. Pour le moment, PSIPRED reste toujours l'un des outils les plus fiables pour la prédiction, 20 ans après. Des évolutions du logiciel ont eu lieu, mais le cœur du fonctionnement est identique.
Cela montre aussi qu'avec un travail sérieux en amont fait sur le traitement des données, sur l'étalonnage de la méthode, tout outil basé sur l'apprentissage peut avoir une fiabilité pérenne.
Références
[1] ANFINSEN CB, « Principles that govern the folding of protein chains », Science. 181 (4096): 223–230, 1973.
[2] LEVINTHAL C., « How to Fold Graciously », Mossbauer Spectroscopy in Biological Systems: Proceedings of a meeting held at Allerton House, Monticello, Illinois: 22–24, 1969
[3] Portail des structures tridimensionnelles de macromolécules : https://www.ebi.ac.uk/pdbe/
[4] Classification des éléments de structure secondaire des protéines : http://www.cathdb.info/
[5] Structure des protéines : https://fr.wikipedia.org/wiki/Structure_des_protéines
[6] Détail de l'algorithme DSSP : https://fr.wikipedia.org/wiki/Structure_secondaire
[7] Les zones phi/psi accessibles en pratique : https://fr.wikipedia.org/wiki/Diagramme_de_Ramachandran
[8] Les premières approches analyses statistiques des éléments de structure secondaire : https://en.wikipedia.org/wiki/Chou%E2%80%93Fasman_method
[9] Une liste non exhaustive des méthodes de prédiction de structure secondaire : https://en.wikipedia.org/wiki/List_of_protein_secondary_structure_prediction_programs
[10] Une très bonne introduction au machine learning (et plus ) : http://www.xavierdupre.fr/app/mlstatpy/helpsphinx/c_ml/rn/rn.html
[11] Utiliser un jeu sérieux pour prédire la structure d'une protéine : https://fold.it/
Pour aller plus loin
Nous avons abordé dans cet article un élément important pour la construction d'un modèle tridimensionnel d'une protéine à partir de sa séquence, qui est l'enjeu de la bio-informatique structurale. Bien que la méthode présentée soit l'une des plus robustes disponible, cette prédiction ne suffit pas en elle seule à répondre à la problématique globale. Il faut ainsi agréger différentes prédictions à plusieurs échelles pour arriver à un modèle fiable. Cela demande beaucoup de temps et d'expertise, mais il est possible de les découvrir pas à pas sous forme de jeu avec FoldIt [11]. Vous pourrez ainsi compléter les concepts introduits dans cet article, puis peut-être participer à des expériences virtuelles utiles pour la recherche contre le cancer !