Pour faire face aux défis de la biologie, la bioinformatique propose des modules qui permettent de rendre accessibles des fonctions basiques et avancées d’analyse de données. Les données traitées sont en général des séquences d'ADN qu’il s’agira d’identifier, d’aligner et d'analyser grâce à la littérature scientifique. Les formats les plus utilisés en bioanalyse, comme les formats FASTA et GenBank demeurent par certains aspects difficiles à manipuler. Nous vous proposons un didacticiel qui introduira les fonctions de Biopython, un module Python dédié à la manipulation de données biologiques dans le cadre de l’analyse complète d’une séquence d’ADN.
« Biology has become an information science ». L'apparition et l'évolution des technologies de séquençage ont accompagné de nouveaux besoins de traitement et d'analyse de données qui sont massives, complexes, interdépendantes et distribuées.
Notre histoire commence comme beaucoup par un mystère… Une séquence ADN de quelques centaines de paires de bases qu’il va falloir traduire, identifier, replacer dans son contexte et analyser en cherchant la littérature scientifique associée. Le séquenceur a produit la séquence suivante :
>>> mysterious_sequence
TCAGACCGTTCATACAGAATTGGCGATCGTTCGGCGTATCGCCGAAATCACCGCCGTAAGCCGACCAGGGGTTGCCGTTA
TCATCATATTTAATCAGCGACTGATCCACGCAGTCCCAGACGAAGCCGCCCTGTAAACGGGGATACTGACGAAACGCCTG
CCAGTATTTAGCGAAACCGCCAAGACTGTTACCCATCGCGTGGGCGTATTCGCAAAGGATCAGCGGGCGCGTCTCTCCAG
GTAGCGATAGCCAATTTTTGATGGACCATTTCGGCACAGCCGGTAAGGGCTGGTCTTCTTCCACGCGCGCGTACATCGGG
CAAATAATTTCGGTGGCCGTGGTGTCGGCTCCGCCGCCTTCATACTGCACCGGGCGGGAAGGATCGACAGATTTGATCCA
GCGATACAGCGCGTCGTGATTAGCGCCGTGGCCTGATTCATTCCCCAGCG
Oui, Biopython s'écrit avec un « p » minuscule, contrairement aux autres (BioPerl, BioJava, etc.).
Biopython [1] est un ensemble d’outils écrits en Python pour la biologie computationnelle et la bioinformatique. Il existe des initiatives similaires pour la plupart des langages classiques : BioPerl, BioJava, BioRuby, etc. Les principales fonctionnalités portent sur les manipulations de séquences, la récupération et la manipulation de données depuis des sources classiques comme ExPASy pour les données de génomique ou de protéomique, ou PubMed pour les articles scientifiques.
La principale force de Biopython sont ses « parsers », des modules capables de lire et manipuler les formats standards de données biologiques les plus répandus. La possibilité d’accéder automatiquement aux bases de données en ligne et d’en utiliser les outils permet d’intégrer facilement des traitements dans des workflows d’analyse automatisés. En outre, la documentation en ligne est particulièrement riche. La plupart des fonctions sont détaillées dans le Livre de Recettes Biopython [2] et les forums avoisinants.
1. Comment l'ADN est transformé en protéines ?
1.1 De l'ADN à l'ARN
L’ADN est généralement constitué de deux brins enroulés en double hélice. Chaque brin est composé d’une succession de nucléotides composés chacun d’un sucre à cinq atomes de carbone sur lequel sont fixés un phosphate et une des quatre bases azotées : l’adénine (A), la thymine (T), la cytosine (C), ou la guanine (G). Le phosphate de chaque nucléotide est relié au sucre du nucléotide suivant, ce qui définit l’orientation de la lecture de chaque brin, de l’extrémité notée 5’ vers l’extrémité notée 3’.
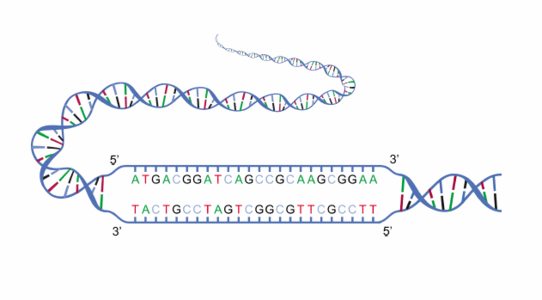
Chaque nucléotide d’un brin trouve son nucléotide correspondant dans l’autre brin : l’adénine s’associant à la thymine et la cytosine à la guanine. Ainsi, les deux brins d’ADN sont dits complémentaires. Les brins sont disposés tête-bêche, c’est-à-dire que l’extrémité 5’ d’un brin est en face de l’extrémité 3’ de l’autre brin. La figure 1 présente les deux brins complémentaires d'un fragment d'ADN. La taille du génome en nucléotides présente une grande variabilité dans la nature, allant de quelques milliers à quelques centaines de milliards de nucléotides en fonction des organismes. Il est difficile de relier la taille du génome au niveau de complexité de l’organisme, mais les organismes procaryotes (par exemple les bactéries) ont tendance à avoir des génomes plus réduits avec un rapport des régions codantes et intergéniques plus élevé que chez les organismes eucaryotes (par exemple les mammifères).
Fig. 1 : Fragments de deux brins complémentaires d’ADN. La lecture de chaque brin se fait de l’extrémité 5’ vers l’extrémité 3’ (adapté de : http://commons.wikimedia.org/wiki/File:DNA_strands.gif).
Chaque brin d’ADN peut être séparé en deux composantes : une fraction codante correspondant aux gènes et dont la séquence détermine une séquence d’acides aminés formant une protéine, et une fraction non codante ou intergénique, dont la fonction est variable, voire inconnue. En fonction des espèces, la taille des génomes varie de quelques centaines à quelques dizaines de milliers de gènes, là encore sans lien marqué avec la complexité des organismes.
Pour qu’un gène soit traduit en protéine, le gène et son environnement proche doivent d’abord être transcrits en ARN. Il s'agit globalement de la simple copie d'un brin d'ADN dans lequel les thymines (T) sont remplacées par des uraciles (U), et le sucre des nucléotides porte un groupement -OH en plus. L’ARN, dit alors « messager », est ensuite pris en charge par des structures complexes appelées les ribosomes, qui vont lire la séquence des nucléotides et produire la protéine correspondante.
1.2 De l'ARN aux protéines
Chaque protéine est donc composée d’une succession d’acides aminés d’une taille variant de quelques dizaines à quelques dizaines de milliers de bases. Il n’existe que 22 types d’acides aminés entrant dans la composition des protéines. Une suite de trois nucléotides, que l’on appelle un codon, suffit donc à coder chaque acide aminé : il y a 43=64 combinaisons possibles par codon, alors qu’une suite de deux nucléotides n’en offre que 42=16. Puisqu’il y a 64 codons pour 22 acides aminés, il y a une certaine redondance et plusieurs codons peuvent conduire au même acide aminé.
Il y a également deux types de codons particuliers : start et stop, qui indiquent respectivement le début et la fin de la région à traduire. Le codon start correspond le plus souvent chez les procaryotes à la méthionine (ATG). La lecture de l’un des codons stop (par exemple TAA) provoque la séparation des sous-unités du ribosome et met fin à la traduction. Le tableau suivant décrit le code génétique et peut être récupéré via Biopython.
>>> from Bio.Data import CodonTable
>>> # correspondance entre les 64 codons et les 22 acides aminés, désignés
... # par une lettre (par exemple « M » désigne la méthionine) ;
... # « (s) » désigne un codon start..
...
>>> print(CodonTable.unambiguous_dna_by_id[1])
| .T. | .C. | .A. | .G. |
–---+---------+---------+---------+---------+----
T.. | TTT F | TCT S | TAT Y | TGT C | ..T
T.. | TTC F | TCC S | TAC Y | TGC C | ..C
T.. | TTA L | TCA S | TAA Stop| TGA Stop| ..A
T.. | TTG L(s)| TCG S | TAG Stop| TGG W | ..G
–---+---------+---------+---------+---------+----
C.. | CTT L | CCT P | CAT H | CGT R | ..T
C.. | CTC L | CCC P | CAC H | CGC R | ..C
C.. | CTA L | CCA P | CAA Q | CGA R | ..A
C.. | CTG L(s)| CCG P | CAG Q | CGG R | ..G
–---+---------+---------+---------+---------+----
A.. | ATT I | ACT T | AAT N | AGT S | ..T
A.. | ATC I | ACC T | AAC N | AGC S | ..C
A.. | ATA I | ACA T | AAA K | AGA R | ..A
A.. | ATG M(s)| ACG T | AAG K | AGG R | ..G
–---+---------+---------+---------+---------+----
G.. | GTT V | GCT A | GAT D | GGT G | ..T
G.. | GTC V | GCC A | GAC D | GGC G | ..C
G.. | GTA V | GCA A | GAA E | GGA G | ..A
G.. | GTG V | GCG A | GAG E | GGG G | ..G
–---+---------+---------+---------+---------+----
1.3 Les six cadres de lecture
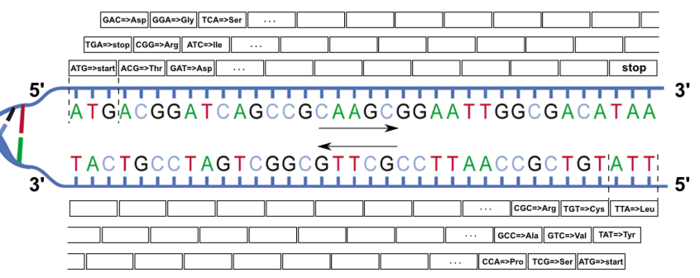
À partir d’une séquence brute d’ADN, identifier les gènes demande de repérer les fractions codantes, c’est-à-dire les portions d’ADN comprises entre un codon start et un codon stop. Cela soulève deux difficultés : d’une part on ne sait pas sur quel brin se fera la traduction en protéine, et d’autre part on ne sait pas par quel nucléotide commence le premier codon.
À partir d’une séquence d’ADN, on a donc six cadres de lecture à analyser : trois sur la séquence en se décalant à chaque fois d’un nucléotide de l’extrémité 5’ vers l’extrémité 3’, et trois autres sur la séquence du brin complémentaire en allant, là encore, de 5’ vers 3’. La figure 2 donne un exemple des six cadres de lecture possibles à partir du fragment d’ADN de la figure 1.
À quelques exceptions près, notamment chez les virus, les gènes ne sont pas chevauchants, aussi un seul des six cadres de lecture est susceptible de correspondre à un gène.
Fig. 2 : Les six cadres de lecture pour un fragment d'ADN (adapté de http://commons.wikimedia.org/wiki/File:DNA_ORF.gif)
2. Traiter les séquences biologiques avec Biopython
On pourrait bien sûr écrire le code pour lire la séquence d’ADN à partir du fichier FASTA (voir encadré), rechercher un gène dans les six cadres de lecture et le traduire en séquence d’acides aminés, mais il s’agit-là d’opérations usuelles en bioinformatique et il est bien plus simple d’utiliser des librairies comme Biopython.
Les formats FASTA, FASTQ et GenBank
FASTA, FASTAQ et GenBank sont trois formats de fichiers textes employés en bioinformatique.
FASTA permet de stocker des séquences en les faisant précéder d'un commentaire :
> Commentaire
AGTCGAATG...
Le format FASTAQ est une évolution du format FASTA, qui ajoute des scores de qualité aux données FASTA (pourcentage d'identification correcte d'une base). Ce format est utilisé par les séquenceurs à haut débit.
Enfin, le format GenBank est le format utilisé par la banque de données du même nom et qui fournit des informations complémentaires sur les séquences. Ici, chaque ligne contient un mot-clé indiquant à quoi correspond la valeur qui suit sur la ou les ligne(s) suivante(s). Exemple :
LOCUS SCU49845 5028 bp DNA PLN 21-JUN-1999
DEFINITION Saccharomyces cerevisiae TCP1-beta gene, partial cds, and Axl2p
(AXL2) and Rev7p (REV7) genes, complete cds.
...
Biopython s'installe comme beaucoup de modules Python par :
$ pip install biopython
2.1 Importer une séquence : objets BioSeq et BioSeqIO
Dans Biopython, les séquences sont des instances de la classe Bio.Seq. Elles peuvent être importées de deux façons différentes. Lorsqu’on a simplement une séquence au format texte, on importe la classe Seq, puis on crée une instance de l’objet. Le second paramètre du constructeur de Seq permet d’indiquer l’alphabet, afin de distinguer une séquence protéique d’une séquence nucléotidique.
from Bio.Seq import Seq
from Bio.Alphabet import generic_dna, generic_protein
myNucleotidicSequence = Seq('TCAGACCG[...]CCAGCG', generic_dna)
# ou
myProteicSequence = Seq('NYLLSH[...]IYTH', generic_protein)
Lorsque la séquence est contenue dans un fichier au format FASTA, il est possible de l’importer directement depuis le fichier :
from Bio import SeqIO
from Bio.Alphabet import generic_dna
mysterious_sequence = SeqIO.read(open('my_sequence.fasta'),
'fasta',
alphabet=generic_dna).seq
Le module SeqIO génère des objets SeqRecord, lesquels contiennent entre autres une séquence (.seq) et un identifiant (.id). SeqIO est capable de charger plusieurs formats de fichier (e.g. FASTA, FASTQ, GenBank) et se révèle capable de lire un fichier contenant plusieurs entrées d’un même format indiqué par le second paramètre de parse(), par exemple genbank, fasta ou fastq :
from Bio import SeqIO
my_genbank_records = []
for gbk_record in SeqIO.parse(open('mon_fichier.gbk'),'genbank'):
my_genbank_records.append(gbk_record)
2.2 Manipulations de bases
Une instance de Bio.Seq se comporte comme une chaîne de caractères Python. On peut notamment en extraire des sous-séquences qui sont toujours des instances de Bio.Seq avec les opérateurs classiques : les indices sont numérotés à partir de zéro, et pour la tranche [2:5], le premier élément (celui d’indice 2, donc 'A' pour notre séquence) est inclus et le dernier élément (celui d’indice 5 ci-dessous, donc 'C') est exclu. Les indices de début et de fin sont optionnels. Dans ce cas, on commence au début de la chaîne ou on va jusqu’à sa fin.
print(mysterious_sequence) # renvoie 'TCAGACCGTTCAT...'
str(len(mysterious_sequence)) # renvoie 450
# (la longueur de la séquence)
print(mysterious_sequence[2:5]) # renvoie 'AGA'
# (de l'indice 2 inclus
# à l'indice 5 exclu)
print(mysterious_sequence[:5]) # renvoie 'TCAGA'
#(du début à l’indice 5 exclu)
print(mysterious_sequence[2:]) # renvoie 'AGACCGTTCAT[…]'
# (de l’indice 2 inclus à la fin)
Enfin, en utilisant un pas de trois et en faisant varier l’indice de début, on peut obtenir séparément les trois bases de chaque codon, ou la première base de chaque codon pour les trois cadres de lecture. En prenant un pas de -1, on obtient la séquence lue en sens inverse (ce qui n’est pas la même chose que le brin complémentaire) :
print(mysterious_sequence[0::3]) # renvoie 'TGCTTAA[…]'
# (indices 0, 3, 6, etc.)
print(mysterious_sequence[1::3]) # renvoie 'CAGCAGT[…]'
# (indices 1, 4, 7, etc.)
print(mysterious_sequence[2::3]) # renvoie 'ACTACAT[…]'
# (indices 2, 5, 8, etc.)
print(mysterious_sequence[::-1]) # renvoie la séquence inversée
# 'GCGACCC[...]CTTGCCAGACT'
La classe Bio.Seq permet d’exploiter le sens biologique des séquences. En partant d’une instance Seq, il est facilement possible d’obtenir la séquence complémentaire (pour les mettre en regard l’une de l’autre, cf. Fig. 1), ou son reverse_complement (qui, du coup, est bien orienté de 5’ vers 3’). Attention : les versions reverse ou complement d’une séquence n’ont aucun sens biologique dans la mesure où l’ADN et l'ARN ne sont lus que dans le sens 5’ -> 3’ quel que soit le brin. Si une séquence trouve des séquences homologues lors d’un alignement type BLAST, ses versions reverse ou complement n’ont aucune raison de trouver les mêmes homologues, contrairement à la version reverse_complement.
brin1 = Seq('CATGTCATAA', generic_dna)
print(brin1) # renvoie (sans surprise) 'CATGTCATAA'
print(brin1.complement()) # renvoie 'GTACAGTATT'
print(brin1 + '\n' + brin1.complement())
brin2 = brin1.reverse_complement()
print(brin2) # renvoie bien 'TTATGACATG' qui correspond au brin complémentaire
# du brin 1, lu dans le sens 5' → 3'
De même, Biopython est capable de traduire une séquence nucléotidique en remplaçant chaque codon par la protéine correspondante (cf. tableau de la section 1.2).
gene = Seq('ATGTCATAA', generic_dna)
print(gene.translate()) # renvoie 'MS*' car le premier codon (ATG)
# correspond à la méthionine (codon start),
# le second codon (TCA) correspond à la
# sérine, et le troisième codon (TAA)
# est un codon stop.
Cependant, Biopython ne traduira la séquence que dans le premier cadre de lecture. Pour récupérer les autres cadres de lecture, il suffira de décaler manuellement la séquence et/ou de prendre son reverse_complement.
print(brin1) # renvoie 'CATGTCATAA'
print(brin1.translate()) # renvoie 'HVI' (H=histidine, V=valine, I=isoleucine)
#
# cadre de lecture +1 et traduction
print(brin1[1:]) # renvoie 'ATGTCATAA'
print(brin1[1:].translate()) # renvoie 'MS*' (M=méthionine/start, S=sérine, *=stop)
#
# cadre de lecture +2 et traduction
print(brin1[2:]) # renvoie 'TGTCATAA'
print(brin1[2:].translate()) # renvoie 'CH' (C=cystéine, H=histidine)
#
# cadre de lecture -1 et traduction
print(brin2) # renvoie 'TTATGACATG'
print(brin2.translate()) # renvoie 'L*H' (L=leucine, *=stop, H=histidine)
#
# cadre de lecture -2 et traduction
print(brin2[1:]) # renvoie 'TATGACATG'
print(brin2[1:].translate()) # renvoie 'YDM' (Y=tyrosine, D=acide aspartique, M=méthionine/start)
#
# cadre de lecture -3 et traduction
print(brin2[2:]) # renvoie 'ATGACATG'
print(brin2[1:].translate()) # renvoie 'MT' (M=méthionine/start, T=thréonine)
2.3 Outils d'analyse
Le module Bio.SeqUtils propose plusieurs outils pour analyser les séquences nucléotidiques : affichage simultané des six cadres de lecture, calcul du taux de GC, GC skew (voir note), recherche de motifs, etc.
Le taux de GC est la proportion de bases G et C dans une séquence. Un taux important de GC peut donner des informations sur des caractéristiques du génome étudié.
Le GC skew est un calcul se basant sur la proportion de bases G et C pour déterminer le début et la fin d'un brin d'ADN (proportion plus importante de G et C dans ces cas-là).
La méthode six_frame_translations() renvoie une visualisation des 2 brins et des 6 cadres de lecture :
from Bio.SeqUtils import six_frame_translations
print(six_frame_translations(mysterious_sequence)) # renvoie une visualisation des 2 brins
# et des 6 cadres de traduction
# GC_Frame: a:98 t:97 g:125 c:130
# Sequence: tcagaccgtt ... ttccccagcg, 450 nt, 56.67 %GC
#
#
# 1/1
# R P F I Q N W R S F G V S P K S P P * A
# Q T V H T E L A I V R R I A E I T A V S
# S D R S Y R I G D R S A Y R R N H R R K
# tcagaccgttcatacagaattggcgatcgttcggcgtatcgccgaaatcaccgccgtaag 53 %
# agtctggcaagtatgtcttaaccgctagcaagccgcatagcggctttagtggcggcattc
# S R E Y L I P S R E A Y R R F * R R L G
# * V T * V S N A I T R R I A S I V A T L
# L G N M C F Q R D N P T D G F D G G Y A
# etc.
Les méthodes GC() et CG123() permettent d’obtenir des taux de GC globaux et/ou par position sur les codons :
from Bio.SeqUtils import GC, GC123
print(GC(mysterious_sequence)) # renvoie 56.666 : le taux de GC pour l’ensemble de la séquence
print(GC123(mysterious_sequence)) # renvoie (56.66, 59.33, 64.0, 46.66) : un tuple contenant
# le taux de GC pour l’ensemble de la séquence,
# puis sur les positions 1, 2 et 3 de chaque codon.
La méthode GC_skew() calcule le ratio (nombre de G – nombre de C) / (nombre de G + nombre de C) par fenêtre de 100 paires de bases. L’évolution de ce ratio permet de localiser l’origine de réplication dans les génomes circulaires.
from Bio.SeqUtils import GC_skew
GC_skew(mysterious_sequence, window=100) # renvoie une liste de ratios (nb G - nb C) / (nb G + nb C)
# par fenêtre de 100 pb. Pour un génome complet,
# le GC skew permet de localiser l’origine de réplication.
Enfin, la méthode nt_search() est particulièrement utile pour rechercher des motifs pouvant contenir des ambiguïtés IUPAC (ambiguïtés sur la détermination des nucléotides).
from Bio.SeqUtils import nt_search
print(nt_search(str(mysterious_sequence),”motif”) # le motif pouvant contenir des ambiguïtés IUPAC
# (e.g. Y = C ou T, N = A, C, T ou G),
# renvoie une liste contenant le motif sans
# ambiguïté ainsi que les positions sur la
# séquence où le motif est trouvé
3. Comparer une séquence avec les banques en ligne
3.1 Lancer des requêtes BLAST en ligne
Il est également possible de lancer des requêtes BLAST (voir note) sur les serveurs du National Center for Biotechnology Information (NCBI). Nous allons nous servir de ce module pour tenter d’identifier notre séquence mystérieuse.
BLAST, pour Basic Local Alignment Search Tool, est un outil permettant d'effectuer des comparaisons entre plusieurs séquences et de retrouver des régions similaires. Cela permet par exemple de prédire la fonction de certaines séquences non annotées en les comparant à des séquences étudiées in vivo.
from Bio.Blast import NCBIWWW
blast_result = NCBIWWW.qblast('blastx', 'nr', mysterious_sequence)
Le résultat BLAST est généré dans le format XML pour être sauvegardé dans un fichier :
with open('my_blast_result.xml', 'w') as result_file:
result_file.write(blast_result.read())
|
E-value |
Accession |
Hit |
% identity |
|
1.47E-98 |
NZ_AKBV01000001.1 |
E. Coli (Lac Z) Beta-Galactosidase |
96.0 % |
|
2.16E-98 |
CAB93483 |
beta-galactosidase [Cloning vector pBRINT-TsCm] |
96.0 % |
|
6.22E-98 |
WP_005148809 |
beta galactosidase small chain family protein [Shigella sonnei] |
96.0 % |
|
6.92E-98 |
WP_004220383 |
beta-galactosidase [Klebsiella pneumoniae] |
96.0 % |
3.2 Récupérer les génomes des meilleurs candidats
Les résultats BLAST indiquent que la séquence mystérieuse est très proche du génome de la bactérie Escherichia coliet plus particulièrement du gène lacZ, codant pour une β-galactosidase (enzyme).
Nous allons récupérer les génomes de deux organismes proches et en extraire le gène lacZ. Le module Entrez permet d’accéder aux bases de données du NCBI pour lancer une recherche ou télécharger une séquence.
from Bio import Entrez
Entrez.email = 'A.N.Other@example.com' # Il est important de signaler au NCBI qui lance des requêtes
handle = Entrez.esearch(db="nucleotide", term="Escherichia[Orgn] AND lacZ[Gene]")
record = Entrez.read(handle)
record["IdList"] # renvoie une liste de 20 identifiants de séquence
# répondant aux critères de la recherche
Pour récupérer les données :
from Bio import Entrez
Entrez.email = "A.N.Other@example.com"
download = Entrez.efetch(db='nucleotide',
id='NZ_AKBV01000001.1',
rettype='gbwithparts',
retmode='text')
with open('NZ_AKBV01000001.1.gbk','w') as outfile:
outfile.write(str(download.read())) # génère un fichier contenant le fichier téléchargé
Le format GenBank est un format riche permettant de stocker des séquences et des annotations. Il est en général constitué d’un en-tête, qui décrit l’organisme dont provient la séquence, d’un corps, qui contient l’ensemble des annotations (source, gènes, CDS, ARNribosomiques, etc.) et un bloc 'ORIGIN' qui contient la séquence complète. Cette richesse d’informations le rend assez difficile à manipuler.
Biopython possède un parser pour les fichiers au format GenBank et génère des objets record.
from Bio import SeqIO
with open('my_file.gbk') as gbk_file:
record = SeqIO.read(gbk_file,'genbank')
Il est ensuite possible d’itérer sur les différentes annotations (features) pour récupérer une annotation d’intérêt. En général, les trois principaux types d’annotations sont la 'source' qui décrit la séquence complète, les gènes, et les CDS (CoDing Sequence). Les annotations de type CDS ont plusieurs attributs définissant leur type (feature.type), leur position (feature.location), laquelle inclut les bornes et l’orientation de l’annotation, et enfin des descripteurs (feature.qualifiers). Ce dernier attribut correspond à un dictionnaire dont les valeurs sont des dictionnaires contenant l’identifiant, le nom du produit et sa séquence protéique, ou encore la fonction du gène, ainsi que d’éventuelles notes contenant des informations complémentaires.
Il faut noter d’une part que les feature.qualifiers[‘xxx’] sont des listes, même si elles ne contiennent qu’un seul élément, et par ailleurs que les descripteurs ne sont pas obligatoirement présents dans le fichier GenBank, ce qui peut aboutir à des erreurs dans l’utilisation de ces commandes. Aussi, il est recommandé d’utiliser un try.
for feature in record.features:
print feature.type # renvoie par exemple 'source', 'gene' ou 'CDS'
print feature.location # renvoie les bornes et l’orientation de l’annotation
if feature.type == 'CDS':
try:
print feature.qualifiers['gene'] # renvoie le nom du gène
print feature.qualifiers['roduct'] # renvoie le nom de produit pour le gène/CDS
print feature.qualifiers['translation'] # renvoie si elle est précisée la séquence protéique
print feature.qualifiers['function'] # renvoie une description de la fonction du gène/CDS
print feature.qualifiers['notes'] # renvoie une liste de descripteurs supplémentaires
3.3 Aligner des séquences et déterminer l'espèce
Biopython permet également d’utiliser des logiciels d’alignement de séquence comme MUSCLE. Ce module utilise un fichier au format FASTA contenant les séquences à aligner.
from Bio.Align.Applications import MuscleCommandline
from StringIO import StringIO
from Bio import AlignIO
muscle_cline = MuscleCommandline(input='my_sequences_to_align.fasta',out='alignment.aln') #ligne de commande
stdout, stderr = muscle_cline()
4. Interroger la banque bibliographique PubMed
Biopython a permis de comparer notre séquence mystère avec d’autres séquences connues et regroupées dans GenBank, afin de déterminer de quel(s) gène(s) elle est la plus proche et d’en identifier l’espèce. Il permet également d’accéder à de nombreuses informations relatives à ce(s) gène(s).
Comme à la section 3.2, ces informations sont notamment disponibles via Entrez [3], le système global de recherche inter-bases du Centre Américain pour les Informations Biotechnologiques (NCBI). Il permet notamment de faire des requêtes assez fines en combinant les données d’une quarantaine de bases dont notamment Gene sur les gènes (leurs séquences, les voies métaboliques dans lesquelles ils interviennent...), et PubMed sur les articles scientifiques. Entrez est accessible via des formulaires sur des pages web pour une utilisation ponctuelle, ainsi que via eUtilities [4] pour une utilisation par des programmes.
Les eUtilities sont en fait composées de plusieurs outils : eSearch permet de faire une requête sur une base et de récupérer la liste des identifiants des éléments qui correspondent à la requête (par exemple une liste de gènes de la base Gene) ; eLink fonctionne sur le même principe, mais sert à interroger des relations entre les bases (par exemple les articles de la base PubMed qui correspondent à un gène de Gene). Dans les deux cas, eSummary et eFetch permettent de récupérer le résumé ou l’intégralité des éléments à la place de leurs identifiants. Biopython fournit une interface pratique permettant d’interroger ces bases et surtout, de traiter les résultats en s’abstrayant des formats de données. Il prend en charge la communication avec l’interface d’Entrez en respectant le guide d’utilisation [5] (s’identifier en donnant son e-mail, ne pas envoyer plus de trois requêtes par seconde, etc.).
Pour illustrer ces possibilités, nous allons utiliser Entrez pour trouver toutes les versions du gène lacZ chez les différentes souches d’E. coli (après tout, une étude sur une autre souche a peut-être révélé des choses intéressantes). Cela nous permettra d’interroger PubMed pour trouver les articles qui s’y rapportent, puis d’analyser les citations entre ces articles.
4.1 Interroger une base d'Entrez : utiliser Gene pour trouver les identifiants de lacZ dans les différentes souches d'Escherichia coli
Interroger une des bases d’Entrez se fait grâce à la fonction esearch() du module Bio.Entrez. Cette fonction prend deux paramètres : le nom de la base dans db, et la requête dans term. La requête est une chaîne de caractères dont la syntaxe est identique à celle générée sur le formulaire. L’appel à esearch envoie la requête. La fonction read() du module Bio.Entrez lit le résultat et le met dans un dictionnaire dont la clé Count indique le nombre de réponses (ici 57), et la clé IdList indique la liste des identifiants (ici des geneID, puisque l’on interroge la base Gene).
Il faut cependant remarquer que par défaut, cette liste est tronquée aux vingt premières valeurs. Il faut donc se servir de la valeur de Count pour faire un second appel à esearch() en précisant le paramètre optionnel retmax. Cette fois-ci, la valeur d’IdList contiendra bien les 57 identifiants.
from Bio import Entrez
Entrez.email = "A.N.Other@example.com"
handle = Entrez.esearch(db="gene", term='(lacZ[sym]) and "Escherichia coli"[orgn]')
record = Entrez.read(handle)
print("Nb candidate gene entries: " + record["Count"])
geneIdList = []
handle = Entrez.esearch(db="gene", term=query, retmax=record["Count"])
record = Entrez.read(handle)
for currentGeneID in record["IdList"]:
geneIdList.append(currentGeneID)
4.2 Interroger les associations entre plusieurs bases d'Entrez : utiliser PubMed pour trouver tous les articles concernant le gène lacZ d'E. coli
Pour interroger PubMed, on pourrait procéder de même qu’avec la base Gene. Cependant, les critères de la requête ne pourront porter que sur les champs de la base PubMed : le titre des articles, la liste de ses auteurs, etc. Si on souhaite trouver les articles qui concernent lacZ chez E. coli, on a en fait besoin d’utiliser le lien qui existe entre Gene et PubMed.
On ne fait donc plus appel à la fonction esearch(), mais à elink() qui prend trois paramètres : dbfrom indique la base à partir de laquelle on fait la requête (ici Gene), db indique la base de laquelle on souhaite obtenir des résultats (ici PubMed) et id indique l’identifiant de la base désignée par dbfrom. Pour obtenir les identifiants des articles relatifs au gène 945006, on fait donc appel à elink(dbfrom='gene', db='pubmed', id='945006').
pmidsForGene = []
for currentGeneID in geneIdList:
handle = Entrez.elink(dbfrom='gene', db='pubmed', id=currentGeneID)
record = Entrez.read(handle)
citations = record[0]['LinkSetDb']
if citations == []:
continue
for currentPMIDdict in citations[0]['Link']:
pmidsForGene.append(currentPMIDdict['Id'])
4.3 Utiliser PubMedCentral pour trouver les citations entre ces articles
Pour trouver les citations entre les articles concernant lacZ, on utilise le même principe : on part de chaque article de PubMed et on va chercher dans PMC la liste des articles qui le citent. Cette fois-ci, dbfrom vaut 'pubmed', db vaut 'pmc' et id prend successivement tous les identifiants de la liste pmidsForGene. Cependant, les résultats de cette requête seront des identifiants dans PMC et non plus dans PubMed. On a donc besoin d’une seconde requête intermédiaire dans l’autre sens pour retrouver l’identifiant dans PubMed (s’il y en a un) correspondant à chaque identifiant PMC.
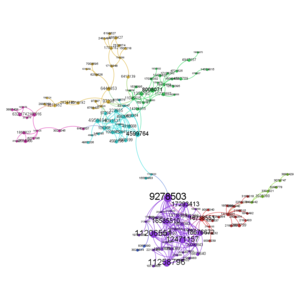
La librairie networkx [6] permet facilement de représenter le graphe dirigé des citations entre articles, et de l’exporter au format GEXF. Gephi [7] permet alors de visualiser le résultat, dont la figure 3 présente la partie principale (cf. [8]). Sans surprise, on retrouve PMID:9278503 parmi les articles les plus cités ; il s’agit de l’article de Science de 1997 annonçant le séquençage complet de la souche K12 d’E. Coli [9].
Fig. 3 : Portion du graphe des articles relatifs au gène lacZ chez Escherichia coli. Chaque sommet est un article désigné par son identifiant dans PubMed. La taille des articles est proportionnelle au nombre de fois où ils sont cités (parmi les autres articles relatifs à lacZ chez E. coli, pas en général).
Conclusion
Biopython permet de réaliser rapidement des tâches classiques de bioinformatique : importer et manipuler des séquences ou des annotations, des alignements, en accédant relativement simplement aux principaux outils et aux principales banques de données, puis en récupérant facilement les résultats.
Pour plus d'informations sur le domaine de la bioinformatique, vous pouvez consulter le blog http://bioinfo-fr.net auquel nous contribuons tous les deux.
Références
[1] Site officiel Biopython : http://biopython.org
[2] Biopython tutorial and cookbook : http://biopython.org/DIST/docs/tutorial/Tutorial.html
[3] Site officiel NCBI Entrez : http://www.ncbi.nlm.nih.gov/gquery/
[4] Site officiel E-utilities quick start : http://www.ncbi.nlm.nih.gov/books/NBK25500/
[5] Site officiel E-utilities usage guidelines and requirements : http://www.ncbi.nlm.nih.gov/books/NBK25497/#chapter2.Usage_Guidelines_and_Requiremen
[6] Site officiel networkx : https://networkx.github.io/
[7] Site officiel Gephi : https://gephi.org/
[8] Blog bioinfo-fr.net : Gephi pour la visualisation et l'analyse de graphes http://bioinfo-fr.net/gephi-pour-la-visualisation-et-lanalyse-de-graphes
[9] The complete genome sequence of Escherichia coli K-12. Blattner FR et al. Science. 1997 5;277(5331):1453-62. http://www.ncbi.nlm.nih.gov/pubmed/9278503